ANS – DIRETRIZ DE UTILIZAÇÃO – DUT 110
DUT 110 – ANÁLISE MOLECULAR DE DNA; PESQUISA DE MICRODELEÇÕES/MICRODUPLICAÇÕES POR FISH (FLUORESCENCE IN SITU HYBRIDIZATION); INSTABILIDADE DE MICROSSATÉLITES (MSI), DETECÇÃO POR PCR, BLOCO DE PARAFINA
1. Cobertura obrigatória quando for solicitado pelo médico assistente (neurologista, oncologista clínico, hematologista ou geneticista) e puder ser realizado em território nacional e for preenchido pelo menos um dos seguintes critérios:
a. na assistência/tratamento/aconselhamento das condições genéticas contempladas nos subitens desta Diretriz de Utilização, quando seguidos os parâmetros definidos em cada subitem para as patologias ou síndromes listadas.
b. na assistência/tratamento/aconselhamento das condições genéticas não contempladas nas Diretrizes dos sub-itens desta Diretriz de Utilização, quando o paciente apresentar sinais clínicos indicativos da doença atual ou história familiar e permanecerem dúvidas acerca do diagnóstico definitivo após a anamnese, o exame físico, a análise de heredograma e exames diagnósticos convencionais.
2. A cobertura de análise molecular de DNA não é obrigatória para as patologias/síndromes, exames ou técnicas listadas a seguir:
a. Ostecondromas hereditários múltiplos (exostoses hereditárias múltiplas); Neurofibromatose 1; Fenilcetonúria; Hipercolesterolemia familiar, Pesquisa dos polimorfismos C677T e A1298 do gene MTHFR e Pesquisa dos polimorfismos 4G/5G no gene PAI1.
b. exames realizados por técnicas de pesquisas em painel, tais como PCR Multiplex, CGH-Array (Hibridização Genômica Comparativa), MLPA (Multiplex Ligation-dependent Probe Amplification), Sequenciamento de Nova Geração (NGS), Sequenciamento completo de todos os éxons do Genoma Humano (Exoma) e Sequenciamento do Genoma (Genoma) humano, nutricional, esportivo, tumoral ou mitocondrial não estão contemplados na letra “b” do item 1. Da mesma forma, o screening de risco pessoal ou screening de planejamento familiar em paciente assintomático, não está contemplado na letra “b” do item 1.”
OBS geral 1: Nas diretrizes de utilização abaixo são considerados:
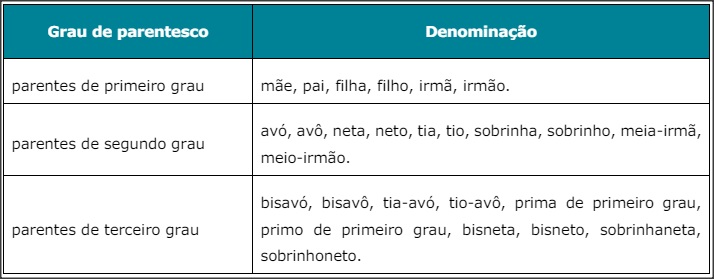
OBS geral 2: Para as diretrizes de utilização em que o método escalonado contemple a técnica CGH-Array (Hibridização Genômica Comparativa), a resolução mínima obrigatória é a densidade de 180k. No caso de plataformas que utilizem apenas SNP- array (Polimorfismo de um único nucleotídeo), a resolução mínima obrigatória é a densidade de 750k.
OBS geral 3: O sequenciamento por NGS dos genes associados a cada síndrome tanto para genes individuais quanto para paineis deve ser realizado na região codificadora do gene e se estender também às regiões intrônicas adjacentes aos exons (pelo menos seis, idealmente dez nucleotídeos imediatamente adjacentes às extremidades 5′ e 3′ dos exons). A análise deve ser realizada de forma a contemplar 100% da extensão da sequencia codificadora e junções intron/exon de todos os genes com cobertura mínima de 20X em todas as regiões analisadas. Em relação a análise de rearranjos quando analisada por NGS o laboratório deve apresentar evidência de validação do protocolo contra outra metodologia de detecção de rearranjos (MLPA ou Array), descrevendo a sensibilidade e os limites de detecção (tamanho de rearranjos) do método.
OBS geral 4: O sequenciamento por NGS dos genes associados a cada síndrome tanto para genes individuais quanto para painéis deve ser realizado na região codificadora do gene e se estender também às regiões intrônicas adjacentes aos exons (pelo menos seis, idealmente dez nucleotídeos imediatamente adjacentes às extremidades 5′ e 3′ dos exons). É recomendado que análise seja realizada de forma a contemplar 100% da extensão da sequencia codificadora e junções intron/exon de todos os genes analisados (teste tipo A) com cobertura mínima de 20X em todas as regiões analisadas. Se a cobertura do painel NGS for inferior a 100%, recomenda-se que regiões não cobertas pelo painel NGS sejam analisadas por outra metodologia (p.ex. sequenciamento de Sanger) de forma a atender a definição de teste tipo A. Caso não tenham sido, deve ser descrito de forma clara o porcentual e número de bases não cobertas, e feita uma observação que o teste não exclui variantes patogênicas nessas regiões. O teste deve ser classificado no laudo como tipo A ou tipo B. De acordo com o EuroGentest (Gert Matthijs et al, Guidelines for diagnostic next-generation sequencing. European Journal of Human Genetics. 2016. 24, 2–5; doi:10.1038/ejhg.2015.226), testes com painéis que demonstrem 100% de cobertura de todos os genes incluidos no painel e regiões intrônicas adjacentes deverão ser denominados do “Tipo A”. Testes onde apenas os genes principais (core genes) são completamente sequenciados, e outros genes possam conter áreas não sequenciadas deverão ser denominados “Tipo B”. Os testes onde não se garante a cobertura completa de nenhum gene do painel denominados “Tipo C” em geral não são recomendados para diagnóstico clínico e por isso, não devem ser oferecidos pelas operadoras de planos de saúde e não são considerados para constatação do atendimento da cobertura mínima obrigatória. Essa nomenclatura de classificação do teste genético, em especial quando se trata de análise com painel de genes, deve ser adotada pelos laboratórios executantes.
OBS geral 5: O material inicial a ser utilizado para o sequenciamento é o DNA.
OBS geral 6: Em caso de detecção de deleção de exon único no MLPA este resultado deve ser confirmado com análise independente.
OBS geral 7: Para as diretrizes de oncogenética em que existam múltiplos indivíduos afetados na mesma família, testar inicialmente e preferencialmente o indivíduo mais jovem afetado pelo tumor típico do espectro avaliado. Quando houver uma mutação identificada na família, familiares não afetados por câncer também poderão ser testados, conforme definido em cada uma das diretrizes específicas. No entanto, em todas as situações de teste genético em familiares, a cobertura será obrigatória somente quando o familiar a ser testado for beneficiário de plano de saúde.
OBS geral 8: Testes genéticos preditivos em menores de idade (análise molecular para doença genética em indivíduo sem evidência fenotípica da doença) devem ser realizados apenas quando forem contemplados todos os seguintes itens:
a) a razão principal de realizar um teste preditivo em menor de idade deve ser em benefício direto de saúde do menor. Não estão justificados testes preditivos quando não houver intervenção justificável que modifique a história natural da doença ou situações de risco, enquanto este for ainda menor de idade;
b) na medida do possível, e considerando a idade do menor, deve ser feita uma tentativa de envolve-lo na decisão sobre a realização do teste, e quando possível obter seu consentimento ou assentimento;
c) os pais ou responsáveis legais pelo menor devem participar tanto quanto possível no processo de tomada de decisão acerca da realização do teste genético. Cabe aos pais ou responsáveis fornecer ao menor, na medida do possível, informações sobre a condição genética a ser testada e o risco genético, podendo haver auxílio e suporte de profissionais especializados em aconselhamento genético no processo. Se a decisão dos pais ou responsáveis não for no sentido de beneficiar diretamente o menor, então os profissionais da saúde têm a responsabilidade de defender os interesses do menor no processo;
d) Aconselhamento genético pré- e pós-teste deve ser sempre realizado quando for indicado teste genético preditivo em menores assintomáticos.
Doenças ou síndromes de cobertura obrigatória referentes ao item a desta Diretriz de Utilização:
110.1 – ACONDROPLASIA/HIPOCONDROPLASIA
1. Cobertura obrigatória para pacientes que apresentem baixa estatura desproporcionada quando restarem dúvidas diagnósticas acerca da doença apresentada após a investigação clínica e radiológica e for preenchido pelo menos um dos seguintes critérios:
a. achados clínicos e radiológicos sugestivos de Acondroplasia (macrocrania com fronte ampla e/ou rizomelia e/ou limitação da extensão dos cotovelos e/ou braquidactilia e/ou configuração das mãos em tridente e/ou geno varo e/ ou ossos tubulares curtos e/ou estreitamento da distância interpedicular da coluna espinhal e/ou hiperlordose lombar e/ou ilíacos arredondados e acetábulos horizontalizados e/ou incisura sacro isquiática pequena e/ou radioluscência femural proximal e/ou leves alterações metafisárias);
b. achados clínicos e radiológicos sugestivos de Hipocondroplasia (macrocrania com face relativamente normal e/ou rizomelia e/ou mesomelia e/ou limitação da extensão dos cotovelos e/ou leve frouxidão ligamentar e/ou mãos e dedos curtos e/ou geno varo e/ou hiperlordose lombar e/ou deficiência intelectual e/ou acantose nigricans e/ou epilepsia do lobo temporal e/ou osteoartrite em adultos e/ou encurtamento dos ossos longos com leve alteração metafisária e/ou braquidactilia e/ou estreitamento da distância interpedicular da coluna espinhal e/ou ilíacos encurtados e quadrados e/ou encurtamento do segmento distal da ulna, alongamento do segmento distal da fíbula e/ou teto do acetábulo raso).
Método de análise:
1. Em caso de achados clínicos e radiológicos sugestivos de Acondroplasia, realizar análise apenas das mutações específicas para Acondroplasia c.1138G>A e c.1138G>C no gene FGFR3.
2. Em caso de achados clínicos e radiológicos sugestivos de Hipocondroplasia, realizar análise apenas das mutações específicas para Hipocondroplasia c.1620C>A e c.1620C>G no gene FGFR3.
110.2 – ADRENOLEUCODISTROFIA
1. Cobertura obrigatória para pacientes do sexo masculino com manifestações clínicas (forma cerebral infantil, adolescente e do adulto, adrenomieloneuropatia e doença de Addison) e diagnóstico bioquímico (dosagem de ácidos graxos de cadeia muito longa).
2. Cobertura obrigatória para pacientes do sexo feminino com manifestações clínicas de adrenomieloneuropatia com diagnóstico bioquímico (dosagem de ácidos graxos de cadeia muito longa) inconclusivo.
3. Cobertura obrigatória em crianças do sexo masculino assintomáticas, cuja mãe possua diagnóstico molecular confirmado de heterozigota para adrenoleucodistrofia.
4. Cobertura obrigatória para o aconselhamento genético de mulheres assintomáticas (parentes de 1º, 2º e 3º graus do caso índice na família), com o diagnóstico molecular de adrenoleucodistrofia no caso índice na família.
Método de análise utilizado de forma escalonada:
1. Nos casos em que a mutação genética já foi identificada na família, realizar apenas a pesquisa da mutação específica.
2. Para os casos não enquadrados no item anterior, realizar Sequenciamento de Nova Geração ou Sequenciamento bidirecional pelo método analítico de Sanger dos éxons do gene ABCD1.
110.3 – AMILOIDOSE FAMILIAR (TTR)
1. Cobertura obrigatória para pacientes de ambos os sexos que apresentem neuropatia autonômica ou sensório-motora lentamente progressiva com biópsia de tecido demonstrando depósito de substância amiloide, especificamente marcados com anticorpos anti-TTR e quando preenchido pelo menos um dos seguintes critérios:
a. bloqueio da condução cardíaca;
b. cardiomiopatia;
c. neuropatia;
d. opacidade do corpo vítreo.
2. Cobertura obrigatória para familiar assintomático de 1º grau ou 2º graus de caso confirmado através de diagnóstico molecular de amiloidose familiar (TTR) no caso índice na família.
Método de análise utilizado de forma escalonada:
1. Nos casos em que a mutação genética já foi identificada na família, realizar apenas a pesquisa da mutação específica.
2. Análise da mutação VAL30MET no gene TTR.
3. Sequenciamento bidirecional pelo método analítico de Sanger dos éxons 2, 3 e 4 do gene TTR.
110.4 – ATAXIA DE FRIEDREICH
1. Cobertura obrigatória para o diagnóstico de pacientes de ambos os sexos com ataxia progressiva e sem padrão de herança familiar autossômica dominante, quando preenchidos pelo menos dois dos seguintes critérios:
a. perda de propriocepção;
b. arreflexia;
c. disartria;
d. liberação piramidal (Babinski);
e. miocardiopatia;
f. alterações eletroneuromiográficas;
g. resistência à insulina ou diabetes;
h. atrofia cerebelar em ressonância nuclear magnética.
Método de análise utilizado de forma escalonada:
1. Pesquisa de mutação dinâmica por expansão de trinucleotídeos GAA no íntron 1 do gene FXN por Reação em Cadeia da Polimerase (PCR) em gel de agarose ou por eletroforese capilar.
110.5 – ATAXIAS ESPINOCEREBELARES (SCA)
1. Cobertura obrigatória para indivíduos sintomáticos com ataxia de marcha associada ou não a outros sinais neurológicos (distonia, neuropatia periférica, parkinsonismo e alterações da movimentação ocular) independente da idade e com história familiar de herança autossômica dominante.
2. Cobertura obrigatória para indivíduos sintomáticos com ataxia de marcha associada ou não a outros sinais neurológicos (distonia, neuropatia periférica, parkinsonismo e alterações da movimentação ocular) independente da idade e sem história familiar desde que preencha todos os seguintes critérios:
a. doença de início insidioso e curso progressivo;
b. início dos sintomas há mais de 6 meses;
c. ressonância magnética de encéfalo que não sugira outra causa para a ataxia (esclerose múltipla, infecção de sistema nervoso central, tumores, mal formações cerebrais e/ou cerebelares, siderose superficial).
3. Cobertura obrigatória para o aconselhamento genético dos familiares de 1º, 2º ou 3º graus assintomáticos maiores de 18 anos quando o diagnóstico molecular do tipo de SCA tiver sido confirmado na família. No caso em que o diagnóstico molecular confirmar SCA do tipo 10 a cobertura para indivíduos assintomáticos não é obrigatória.
Método de análise utilizado de forma escalonada:
1. Nos casos em que a mutação genética já tenha sido identificada na família, realizar apenas a pesquisa da expansão no gene específico.
2. Pesquisa de expansão CAG no gene ATXN3 (SCA3 ou Doença de Machado- Joseph) por Reação em Cadeia da Polimerase (PCR) com análise de fragmentos por eletroforese capilar ou por PCR multiplex fluorescente.
3. Se item anterior for normal, pesquisar simultaneamente expansão de nucleotídeos CAG nos genes ATXN1, ATXN2, CACNA1A, ATXN7, e expansão de pentanucleotídeos ATTCT no gene ATXN10 (SCA1, SCA2, SCA6, SCA7 e SCA10, respectivamente) por Reação em Cadeia da Polimerase (PCR) com análise de fragmentos por eletroforese capilar ou por PCR multiplex fluorescente.
110.6 – ATROFIA MUSCULAR ESPINHAL – AME
1 . Cobertura obrigatória para pacientes de ambos os sexos que apresentem as formas congênitas de Atrofia Muscular Espinhal (artrogripose múltipla congênita ou neuropatia axonal congênita) com hipotonia grave e dependência de suporte respiratório.
2. Cobertura obrigatória para pacientes de ambos os sexos com quadro clínico de atrofia muscular espinhal do tipo I com início dos sintomas antes dos 6 meses de idade, apresentando hipotonia grave, quando preenchidos pelo menos dois dos seguintes critérios:
a. atraso grave do desenvolvimento motor;
b. fasciculação da língua;
c. tremor postural dos dedos;
d. ausência de reflexos tendíneos;
e. ausência de perda sensória.
3. Cobertura obrigatória para pacientes de ambos os sexos com quadro clínico de atrofia muscular espinhal do tipo II com início dos sintomas entre 6 e 18 meses de idade, com hipotonia ou fraqueza muscular progressiva e quando presentes pelo menos dois dos seguintes critérios:
a. fasciculação da língua;
b. tremor postural dos dedos;
c. ausência de reflexos tendíneos;
d. ausência de perda sensória.
4. Cobertura obrigatória para pacientes de ambos os sexos com quadro clínico de atrofia muscular espinhal do tipo III com início dos sintomas após 18 meses de idade com fraqueza muscular progressiva, quando presentes pelo menos dois dos seguintes critérios:
a. fraqueza muscular simétrica proximal;
b. fasciculação da língua ou outros grupos musculares;
c. tremor postural dos dedos;
d. hiporreflexia;
e. cãibras.
5. Cobertura obrigatória para pacientes de ambos os sexos com quadro clínico de atrofia muscular espinhal tipo IV com início dos sintomas na vida adulta e que apresentem eletroneuromiografia com denervação e redução da amplitude do potencial de ação motor, quando presentes pelo menos dois dos seguintes critérios:
a. fraqueza muscular simétrica proximal;
b. fasciculação da língua ou outros grupos musculares;
c. tremor postural dos dedos;
d. hiporreflexia;
e. cãibras.
6. Cobertura obrigatória para o aconselhamento genético de indivíduos assintomáticos, com parentes de 1º ou 2º graus com diagnóstico molecular confirmado.
Método de análise utilizado de forma escalonada:
Para pacientes enquadrados nos itens 1, 2, 3, 4 ou 5:
1. Realizar análise da deleção ou conversão do éxon 7 de ambas as cópias do gene SMN1 por Reação em Cadeia da Polimerase (PCR) em gel de agarose ou por eletroforese capilar.
2. Realizar pesquisa de mutação por Sequenciamento de Nova Geração ou Sanger de toda região codificadora do gene SMN1 quando for diagnosticada heterozigose do éxon 7 do gene SMN1 e o paciente preencher pelo menos dois dos seguintes critérios:
a. eletroneuromiografia revelando denervação e redução da amplitude do potencial de ação motor;
b. biópsia muscular com atrofia de fibras do grupo 1 e 2;
c. creatinoquinase em valores normais.
Para pacientes enquadrados no item 6:
1. Realizar nos pais do paciente reação em Cadeia da Polimerase (PCR) em gel de agarose ou por eletroforese capilar para testar a deleção ou conversão do éxon 7 do gene SMN1 para a deleção do éxon 7 do gene SMN1
2. Quando o resultado for negativo para a deleção do éxon 7, testar para a mutação familiar do gene SMN1 já detectada.
110.7 – CÂNCER DE MAMA E OVÁRIO HEREDITÁRIOS – GENE BRCA1/BRCA2
1. Cobertura obrigatória para mulheres com diagnóstico atual ou prévio de câncer de mama quando preenchido pelo menos um dos seguintes critérios:
a. Diagnóstico de câncer de mama em idade ≤ 35 anos;
b. Diagnóstico de câncer de mama em idade ≤ 50 anos e mais um dos seguintes critérios:
I. um segundo tumor primário da mama (*);
II. ≥ 1 familiar de 1º, 2º e 3º graus com câncer de mama e/ou ovário;
c. Diagnóstico de câncer de mama em idade ≤ 60 anos se câncer de mama triplo negativo (Receptor de estrogênio (RE), Receptor de progesterona (RP) e Receptor HER2 negativos);
d. Diagnóstico de câncer de mama em qualquer idade e mais um dos seguintes:
I. ≥ 1 familiar de 1º, 2º e 3º graus com câncer de mama feminino em idade ≤ 50 anos;
II. ≥ 1 familiar de 1º, 2º e 3º graus com câncer de mama masculino em qualquer idade;
III. ≥ 1 familiar de 1º, 2º e 3º graus com câncer de ovário em qualquer idade;
IV. ≥ 2 familiares de 1º, 2º e 3º graus do mesmo lado da família com câncer de mama em qualquer idade;
V. ≥ 2 familiares de 1º, 2º e 3º graus do mesmo lado da família com câncer de pâncreas ou próstata (escore de Gleason > 7) em qualquer idade.
(*) No caso de câncer de mama bilateral ou duas neoplasias primárias na mesma mama (comprovado por laudos anatomo-patológicos), cada um dos tumores deve ser considerado independentemente.
2. Cobertura obrigatória para mulheres com diagnóstico atual ou prévio de câncer de ovário (tumor epitelial) em qualquer idade e independente da história familiar.
3. Cobertura obrigatória para homens com diagnóstico atual ou prévio de câncer de mama em qualquer idade e independente da história familiar.
4. Cobertura obrigatória para pacientes com câncer de pâncreas e ≥ 2 familiares de 1º, 2º e 3º graus do mesmo lado da família com câncer de mama e/ou ovário e/ou pâncreas ou próstata (escore de Gleason ≥ 7) em qualquer idade.
5. Cobertura obrigatória para pacientes com câncer de próstata (escore de Gleason ≥ 7) e ≥ 2 familiares de 1º, 2º e 3º graus do mesmo lado da família com câncer de mama e/ou ovário e/ou pâncreas ou próstata (escore de Gleason ≥ 7) em qualquer idade.
6. Cobertura obrigatória para teste das 3 mutações fundadoras Ashkenazi nos genes BRCA1 e BRCA2 em pacientes de origem judaica Ashkenazi quando preenchido pelo menos um dos seguintes critérios:
a. câncer de mama em qualquer idade e independente da história familiar;
b. câncer de ovário em qualquer idade e independente da história familiar;
c. câncer de pâncreas em qualquer idade com ≥ 1 familiar de 1º, 2º e 3º. Graus com câncer de mama, ovário, pâncreas ou próstata (escore Gleason ≥ 7).
7. Cobertura obrigatória para pacientes maiores de 18 anos, diagnosticados ou não com câncer, independente do sexo, quando houver mutação deletéria em BRCA1 ou BRCA2 em familiar de 1º, 2º e 3º graus.
8. Cobertura obrigatória para indivíduos com câncer de mama isolado, que tenham estrutura familiar limitada. Estrutura familiar limitada é a ausência, em pelo menos um dos ramos (materno ou paterno) da família, de pelo menos 2 mulheres familiares de 1o, 2o ou 3o graus que tenha vivido além dos 45 anos de idade no momento da avaliação. Incluem-se nesta descrição indivíduos que desconhecem dados de sua família biológica.
9. Cobertura obrigatória para indivíduos com câncer de mama, mas com estrutura familiar limitada (ausência de 2 familiares de 1º, 2º ou 3º graus do sexo feminino em uma das linhagens – materna ou paterna – que tenha vivido além dos 45 anos de idade).
Método de análise utilizado de forma escalonada:
1. Nos casos em que a mutação genética já foi identificada na família, realizar apenas a pesquisa da mutação específica. Para pacientes de origem judaica Ashkenazi nos quais a mutação familiar for uma mutação fundadora, está justificada a realização da análise das 3 mutações fundadoras Ashkenazi ao invés da análise somente da mutação familiar pela possibilidade da ocorrência de mais de uma mutação em genes BRCA em famílias Ashkenazi. Se a família for de origem judaica Ashkenazi e a mutação familiar não for uma das 3 mutações fundadoras, ainda assim justifica-se a realização do teste destas 3 mutações além da mutação que sabidamente segrega na família.
2. Nos casos de pacientes elencados nos itens 1, 2, 3, 4, 5, 6 e 8 realizar o exame Sequenciamento de Nova Geração de toda região codificadora de BRCA1 e BRCA2 e MLPA de BRCA1 e BRCA2;
3. Nos casos de pacientes enquadrados no item 6, realizar teste das 3 mutações fundadoras Ashkenazi nos genes BRCA1 e BRCA2, a saber: BRCA1 185delAG (c.66_67delAG, p.Glu23fs), BRCA1 5382insC (c.5263insC, p.Gln1756fs), e BRCA2 6174delT (c.5946delT, p.Ser1982fs). Se nenhuma destas mutações for identificada e outros critérios de elegibilidade forem contemplados conforme descrito nos itens 1, 2, 3, 4, 5, 7 e 8, deve ser realizada a análise seguindo os critérios de análise escalona descrito para cada item.
OBS 1: Pacientes enquadradas nesta diretriz e com sequenciamento e MLPA para BRCA1 e BRCA2 negativos, devem ser referenciadas para Diretriz de Painel de câncer de mama e/ou ovário.
OBS 2: Pacientes enquadradas nesta diretriz e que simultaneamente preencham os critérios da Diretriz de Painel de câncer de mama e/ou ovário podem ser referenciadas diretamente para a Diretriz de Painel de câncer de mama e/ou ovário.
OBS 3: Nos pacientes em que forem encontradas mutações patogênicas ou provavelmente patogênicas nos genes BRCA1 ou BRCA2, mesmo que assintomáticos, a mastectomia e a salpingo-ooforectomia redutoras de risco, bem como a reconstrução das mamas são de cobertura obrigatória da mesma forma que a cobertura prevista para pacientes com diagnóstico de câncer, quando indicado pelo médico assistente. Caso a beneficiária não deseje realizar mastectomia a ressonância magnética das mamas anual é de cobertura obrigatória.
OBS 4: Quando da realização de salpingo-ooforectomia redutora de risco em portadoras de mutação de BRCA1 e/ou BRCA2, a análise patológica dos anexos excisados deve ser realizada minuciosamente seguindo protocolo específico. COLOCAR REFERENCIA
OBS 5: Para fins desta DUT, tumores invasivos e in situ da mama serão considerados igualmente na definição “câncer de mama”. Para fins desta DUT, serão incluídos na definição “câncer de ovário” os tumores epiteliais de ovário, trompas de falópio e tumores primários de peritônio.
110.8 – COMPLEXO DA ESCLEROSE TUBEROSA
1. Cobertura obrigatória para pacientes de ambos os sexos que apresentem Esclerose Tuberosa Possível e preencham um critério do Grupo I ou pelo
menos dois critérios do Grupo II*:
* Para pacientes que apresentem Esclerose Tuberosa Definitiva e que preencham dois critérios do Grupo I ou um critério do Grupo I e dois critérios do Grupo II, a cobertura do diagnóstico molecular não é obrigatória.
Grupo I (Sinais maiores):
a. Angiofibromas (três ou mais) ou placas fibróticas cefálicas (face ou couro cabeludo);
b. Fibromas ungueais (dois ou mais);
c. Manchas hipomelanóticas (três ou mais; ≥ 5 mm de diâmetro);
d. Nevo de tecido conjuntivo (Shagreen patch );
e. Múltiplos hamartomas nodulares de retina;
f. Displasia cortical, incluindo tuberosidades e linhas de migração radial na substância branca cerebral;
g. Nódulo subependimário;
h. Astrocitoma subependimário de células gigantes;
i. Rabdomioma cardíaco;
j. Linfangiomiomatose;
k. Angiomiolipoma renal.
Grupo II (Sinais menores):
a. Múltiplas fossetas espalhadas no esmalte dentário(três ou mais);
b. Fibromas intraorais (2 ou mais);
c. Hamartoma não renal;
d. Mancha acrômica na retina;
e. Lesões de pele em “confete”;
f. Cistos renais múltiplos;
OBS: Quando Linfangiomiomatose e angiomiolipomas renais forem concomitantes eles serão considerados sinal clínico único.
2. Cobertura obrigatória para o aconselhamento genético de indivíduos de ambos os sexos com parentes de 1º, 2º ou 3º graus com diagnóstico
molecular confirmado.
Método de análise utilizado de forma escalonada:
1. Nos casos em que a mutação genética já tenha sido identificada na família, realizar apenas a pesquisa da mutação específica.
2. Realizar Sequenciamento de Nova Geração de toda região codificante dos genes TSC1 e TSC2.
3. Quando não for possível realizar o Sequenciamento de Nova Geração, realizar o Sequenciamento por Sanger do gene TSC2. Se não for diagnosticada
mutação patogênica através do Sequenciamento do gene TSC2, realizar o Sequenciamento por Sanger gene TSC1.
4. Nos casos em que o diagnóstico não for estabelecido através dos itens acima, realizar MLPA (Multiplex Ligation-dependent Probe Amplification)
para o gene TSC2.
5. Nos casos em que o diagnóstico não for estabelecido através do item anterior, realizar MLPA (Multiplex Ligation-dependent Probe Amplification)
para o gene TSC1.
110.9 – DEFICIÊNCIA DE ALFA 1 – ANTITRIPSINA
1. Cobertura obrigatória para pacientes com diagnóstico de doença pulmonar obstrutiva crônica (DPOC) ou doença hepática crônica ou paniculite necrosante ou vasculite com anticorpo anti-citoplasma de neutrófilos positivo (ANCA) ou bronquiectasia, quando preenchido pelo menos um dos seguintes critérios:
a. níveis plasmáticos diminuídos de Alfa-1 Antitripsina;
b. presença de inclusões intra-hepáticas positivas para ácido periódico-schiff (PAS);
c. presença de enfisema localizado em lobos inferiores em radiografia ou tomografia de tórax em pacientes com menos de 45 anos.
Método de análise:
1. Pesquisa das variantes S e Z por Reação em Cadeia da Polimerase (PCR) em gel de agarose ou por eletroforese capilar do gene SERPINA1.
110.10 – DISPLASIA CAMPOMÉLICA
1. Cobertura obrigatória para recém-nascidos e crianças que apresentem displasia óssea e encurtamento de membros, quando preenchido pelo menos um dos seguintes critérios:
a. alterações nos achados clínicos e radiológicos sugestivos (macrocrania com fronte ampla e/ou arqueamento do fêmur ou tíbia e/ou hipoplasia de escápula e/ou hipoplasia de púbis e/ou asas ilíacas estreitas e verticalizadas e/ ou deformidades de mãos e pés e/ou platispondilia cervical e/ou tórax estreito
e/ou hipomineralização do esterno e/ou braquidactilia e/ou sequência de Pierre Robin);
b. sexo reverso ou genitália ambígua.
Método de análise:
1. Sequenciamento bidirecional pelo método analítico de Sanger dos três éxons e das regiões de transição éxon/íntron do gene SOX9.
110.11 – DISTROFIA MIOTÔNICA TIPO I E II
1. Cobertura obrigatória para pacientes com fraqueza muscular ou miotonia que apresente a forma clássica ou tardia, com ou sem história familiar quando preenchido pelo menos um dos seguintes critérios:
a. Alterações eletroneuromiográficas;
b. Alterações eletrocardiográficas;
c. Alterações nos níveis de CK sérica;
d. Intolerância à glicose ou diabetes;
e. Hipogonadismo;
f. Catarata.
2. Cobertura obrigatória para pacientes com fraqueza muscular ou hipotonia grave sugestivos da forma infantil ou congênita, com história materna de Distrofia Miotônica.
3. Cobertura obrigatória para familiar assintomático de 1º grau ou 2º grau de caso confirmado através de diagnóstico molecular.
Método de análise utilizado de forma escalonada:
1. Pesquisa de mutação dinâmica por expansão de trinucleotídeos CTG no íntron 1 do gene DMPK por Reação em Cadeia da Polimerase (PCR) em gel de agarose ou eletroforese capilar ou Método de Southern Blot.
2. No caso de pacientes com a forma clássica ou tardia em que o diagnóstico não tenha sido confirmado através do item acima, realizar pesquisa de mutação dinâmica por expansão de repetições CCTG no íntron 1 do gene ZNF9 por Reação em Cadeia da Polimerase (PCR) em gel de agarose ou eletroforese capilar ou Método de Southern Blot.
110.12 – DISTROFIA MUSCULAR DE DUCHENNE/BECKER
1. Cobertura obrigatória para indivíduos do sexo masculino, sintomáticos (fraqueza muscular proximal com CK total elevada e/ou ENMG [eletroneuromiografia] alterada, com ou sem biópsia muscular), para pesquisar o gene distrofina.
2. Para o aconselhamento genético dos familiares de 1º, 2º ou 3º graus do lado materno e do sexo feminino em risco (possibilidade de ser portadora – doença recessiva ligada ao X), quando preenchido pelo menos um dos seguintes critérios:
a. Quando o caso índice tiver diagnóstico molecular estabelecido;
b. Quando o caso índice for falecido, mas tiver diagnóstico clínico e laboratorial estabelecido, mesmo sem diagnóstico molecular.
Método de análise utilizado de forma escalonada:
Para o item 1:
a. Para pesquisa de deleções ou duplicações: PCR multiplex ou MLPA para éxons do gene DMD. Deleções de um éxon simples devem ser confirmadas por um procedimento independente.
b. Apenas se não esclarecido pelos anteriores, Sequenciamento de Nova Geração ou Sequenciamento Completo bidirecional por Sanger das regiões codificantes de todo o gene DMD.
Para o item 2:
a. No caso da mutação ter sido identificada na família, realizar somente a análise específica desta mutação (deleção, duplicação ou análise do éxon específico).
b. No caso de parente falecido com Distrofia Muscular de Duchenne ou Becker sem análise molecular, a investigação deve ser escalonada conforme descrito a seguir:
I. Pesquisa da mãe portadora obrigatória (mãe do caso índice):
i. Para pesquisa de deleções ou duplicações: PCR multiplex ou MLPA para éxons do gene DMD. Deleções de um éxon simples devem ser confirmadas por um procedimento independente.
ii. Apenas se não esclarecido pelos anteriores, Sequenciamento de Nova Geração ou Sequenciamento Completo bidirecional por Sanger das regiões codificantes de todo o gene DMD.
II. Caso a mãe portadora obrigatória (mãe do caso índice) for falecida, realizar a pesquisa na mulher em risco:
i. Para pesquisa de deleções ou duplicações: PCR multiplex ou MLPA para éxons do gene DMD. Deleções de um éxon simples devem ser confirmadas por um procedimento independente.
ii. Apenas se não esclarecido pelos anteriores, Sequenciamento de Nova Geração ou Sequenciamento Completo bidirecional por Sanger das regiões codificantes de todo o gene DMD.
110.13 – DOENÇA DE HUNTINGTON
1. Cobertura obrigatória para indivíduos sintomáticos com presença de pelo menos 2 (dois) dos seguintes critérios:
a. Coreia progressiva ou distonia;
b. Distúrbios psiquiátricos (mudanças na personalidade ou declínio cognitivo ou depressão) independente da faixa etária;
c. História familiar de coreia progressiva sugestiva de herança autossômica dominante.
2. Cobertura obrigatória para o aconselhamento genético de indivíduos sintomáticos que apresentem familiares de 1º, 2º ou 3º graus com diagnóstico de doença de Huntington confirmados por análise molecular.
3. Cobertura obrigatória para o aconselhamento genético de indivíduos assintomáticos acima de 18 anos, em risco, que apresentem familiares de 1º, 2º ou 3º graus confirmados por análise molecular.
Método de análise utilizado de forma escalonada:
1. Pesquisa de expansões de trinucleotídeos CAG por Reação em Cadeia da Polimerase (PCR) com análise de fragmentos por eletroforese capilar do gene HTT.
110.14 – DOENÇAS RELACIONADAS AO COLÁGENO DO TIPO 2 (COL2A1), INCLUINDO DISPLASIA ESPÔNDILO-EPIFISÁRIA CONGÊNITA, DISPLASIA DE KNIEST, DISPLASIA ESPÔNDILO-EPI-METAFISÁRIA DO TIPO STRUDWICK, DISPLASIA PLATISPONDÍLICA DO TIPO TORRANCE, SÍNDROME DE STICKLER TIPO I
1. Cobertura obrigatória para pacientes de ambos os sexos que apresentem características clínico-radiológicas sugestivas de displasia esquelética por colagenopatia do tipo
2 e baixa estatura desproporcionada (abaixo do percentil 5) com tronco curto quando preenchidos pelo menos 2 critérios do Grupo I e pelo menos 3 critérios do Grupo II.
Grupo I (Critérios clínicos):
a. Alta miopia, acima de 6DP;
b. Olhos grandes com face aplainada;
c. Fenda palatina ou úvula bífida;
d. Perda auditiva.
Grupo II (Critérios radiológicos):
a. Atraso de ossificação da epífise proximal da cabeça femoral e do púbis nos lactentes;
b. Platispondilia com defeitos de ossificação anterior;
c. Hipoplasia de processo odontóide de C2;
d. Graus variados de irregularidade epifisária ou metafisária e de encurtamento dos ossos longos;
e. Atraso de ossificação da pélvis, com tetos acetabulares horizontalizados, achatamento da cabeça femoral e coxa vara;
f. Atraso de ossificação dos ossos do carpo e tarso.
2. Cobertura obrigatória para pacientes de ambos os sexos com até 28 dias de vida que apresentem baixa estatura desproporcionada (abaixo do percentil 5) com tronco curto quando preenchidos pelo menos 4 critérios do Grupo II.
Grupo II (Critérios radiológicos):
a. Atraso de ossificação da epífise proximal da cabeça femoral e do púbis nos lactentes;
b. Platispondilia com defeitos de ossificação anterior;
c. Hipoplasia de processo odontóide de C2;
d. Graus variados de irregularidade epifisária ou metafisária e de encurtamento dos ossos longos;
e. Atraso de ossificação da pélvis, com tetos acetabulares horizontalizados, achatamento da cabeça femoral e coxa vara;
f. Atraso de ossificação dos ossos do carpo e tarso.
3. Cobertura obrigatória para o aconselhamento genético de indivíduos de ambos os sexos com parentes de 1º, 2º ou 3º graus com diagnóstico molecular confirmado.
Método de análise utilizado de forma escalonada:
1. Nos casos em que a mutação genética já tiver sido identificada na família, realizar apenas a pesquisa da mutação específica.
2. Realizar Sequenciamento de Nova Geração ou Sanger de toda região codificante do gene COL2A1.
110.15 – DOENÇAS RELACIONADAS AO COLÁGENO DO TIPO 3 (COL3A1), EHLERS-DANLOS TIPO IV E ANEURISMA AÓRTICO ABDOMINAL FAMILIAL (AAA)
1. Cobertura obrigatória para pacientes de ambos os sexos que apresentem características sugestivas de síndrome de Ehlers-Danlos (EDS) tipo IV, sem deformidades esqueléticas quando preenchido 1 (um) critério do Grupo I e pelo menos 2 (dois) critérios do Grupo II.*
Grupo I:
a. Rotura arterial;
b. Rotura intestinal;
c. Rotura uterina durante gravidez;
d. História familial de EDS tipo IV.
Grupo II:
a. Pele fina e translucente;
b. Dismorfismos faciais característicos (lábios e filtro nasogeniano finos, queixo pequeno, nariz afilado, olhos grandes);
c. Acrogeria;
d. Fístula arteriovenosa em carótida;
e. Hiperextensibilidade de pequenas articulações;
f. Rotura muscular ou tendínea;
g. Varizes precoces;
h. Pneumotórax ou pneumohemotorax;
i. Hematomas espontâneos ou após trauma mínimo;
j. Luxações ou subluxações articulares crônicas;
k. Pés equinovaros;
l. Recessão gengival;
* Para pacientes que preencham dois critérios do Grupo I a cobertura do diagnóstico molecular não é obrigatória.
2. Cobertura obrigatória para parentes de 1º, 2º, e 3º graus de ambos os sexos sem necessidade de quadro clínico, quando já tiver sido identificada mutação no caso índice.
Método de análise utilizado de forma escalonada:
1. Nos casos em que a mutação genética já tenha sido identificada na família, realizar apenas a pesquisa da mutação específica.
2. Realizar Sequenciamento de Nova Geração ou Sequenciamento por Sanger de toda região codificante do gene COL3A1.
110.16 – DOENCAS RELACIONADAS AO GENE FMR1 (SÍNDROME DO X FRÁGIL, SÍNDROME DE ATAXIA/TREMOR ASSOCIADOS AO X FRÁGIL – FXTAS E FALÊNCIA OVARIANA PREMATURA – FOP)
1. Cobertura obrigatória para pacientes de ambos os sexos com deficiência intelectual ou atraso do desenvolvimento neuropsicomotor ou autismo apresentando pelo menos um dos seguintes critérios:
a. História familial positiva de deficiência intelectual na linhagem materna;
b. Características físicas ou comportamentais sugestivas da síndrome do X frágil.
2. Cobertura obrigatória para pacientes do sexo feminino com falência ovariana antes dos 40 anos (prematura) sem causa definida e após realização de cariótipo, dosagem de LH e FSH.
3. Cobertura obrigatória para pacientes de ambos os sexos com mais de 50 anos de idade com quadro de ataxia cerebelar progressiva e tremor de intenção com história familiar positiva de doenças relacionadas ao FMR1 e cujas causas comuns não genéticas de ataxia tenham sido excluídas.
4. Cobertura obrigatória para familiar assintomático de 1º, 2º ou 3º graus de caso confirmado através de diagnóstico molecular.
5. Cobertura obrigatória para familiar assintomático de 1º, 2º ou 3º graus de caso confirmado clinicamente, quando o caso índice for falecido sem confirmação molecular.
Método de análise utilizado de forma escalonada:
1. Pesquisa de mutação dinâmica por expansão de trinucleotídeos CGG no gene FMR1 por Reação em Cadeia da Polimerase (PCR) por polimorfismo de comprimento dos fragmentos de restrição em gel de agarose ou por eletroforese capilar.
2. Em caso de Reação em Cadeia da Polimerase (PCR) sugestivo de mutação completa ou pré-mutação grande, confirmar por Método de Southern blot ou eletroforese capilar.
110.17 – FEBRE FAMILIAR DO MEDITERRÂNEO
1. Cobertura obrigatória para pacientes de ambos os sexos, independente da idade, que preencham pelo menos um critério maior e um critério menor, ou dois critérios maiores.
2. Cobertura obrigatória, para pacientes de ambos os sexos, na ocorrência de amiloidose renal secundária (AA) após os 15 anos de vida.
3. Cobertura obrigatória para pacientes sintomáticos de ambos os sexos, independente da idade, quando houver mutação detectada em familiar de 1º, 2º ou 3º graus.
4. Cobertura obrigatória para indivíduo assintomático de ambos os sexos, independente da idade, quando for detectada a mutação p.Met694Val, em familiar de 1º, 2º ou 3º graus.
Critérios Maiores:
- Episódios de febre recorrente com serosite (peritonite, sinovite e pleurite).
- Amiloidose do tipo AA sem doença predisponente.
- Resposta favorável à colchicina.
Critérios Menores:
- Episódios de febre recorrente sem serosite.
- Eritema similar a erisipela.
- Familiar de primeiro grau com diagnóstico clínico de febre familiar do mediterrâneo.
Método de análise utilizado de forma escalonada:
1. Para os indivíduos enquadrados nas situações 1 e 2 – Sequenciamento completo do gene MEFV, por Sanger ou sequenciamento de nova geração.
2. Para os indivíduos enquadrados na situação 3 – realizar primeiro pesquisa de mutação familiar conhecida do gene MEFV; caso negativa, e apresente critérios para as situações 1 ou 2, proceder com o Sequenciamento completo do gene MEFV, por Sanger ou sequenciamento de nova geração.
3. Para os indivíduos enquadrados na situação 4 – realizar somente a pesquisa da mutação p.Met694Val.b
110.18 – FIBROSE CÍSTICA E DOENÇAS RELACIONADAS AO GENE CFTR
1. Cobertura obrigatória para pacientes de ambos os sexos com manifestações clínicas de Fibrose Cística e pelo menos dois testes bioquímicos duvidosos ou normais realizados em dias diferentes (dosagem de Cloro no suor normais ou limítrofes <60meq/l).
2. Cobertura obrigatória para recém-nascido com teste de triagem neonatal alterado para fibrose cística (hipertripsinemia – IRT) em pelo menos duas dosagens realizadas em dias diferentes.
3. Cobertura obrigatória para pacientes do sexo masculino com cariótipo normal e azoospermia obstrutiva confirmada através de pelo menos dois espermogramas realizados em dias diferentes e exame de imagem que demonstre agenesia de ductos deferentes.
4. Cobertura obrigatória para o aconselhamento genético de indivíduos assintomáticos e sem história de pai ou mãe com Fibrose Cística, quando o parceiro/cônjuge tiver diagnóstico bioquímico ou molecular de Fibrose Cística.
Método de análise utilizado de forma escalonada:
1. Nos casos em que as mutações nos dois alelos do gene CFTR já tiverem sido identificadas na família, realizar apenas a pesquisa destas mutações específicas.
2. Nos casos em que a mutação genética ainda não foi identificada na família, realizar análise da mutação DF508 no gene CFTR.
3. Para os pacientes enquadrados no item 2, caso tenham uma ou nenhuma mutação DF508 realizar Sequenciamento bidirecional pelo método de Sanger ou Sequenciamento de Nova Geração dos éxons do gene CFTR.
4. Para os pacientes enquadrados nos itens 1, 3 ou 4, caso tenham uma ou nenhuma mutação DF508 realizar painel para fibrose cística com pelo menos as seguintes mutações G542X, S549R, G551D, Q552X G1244E, G1349D, G178R, G551S, S1251N, S1255P, S549N, no gene CFTR. No caso do exame anterior ser negativo, realizar painel de pelo menos 32 mutações para o sexo feminino e 32 mutações e pesquisa variantes poliT no intron 8 para o sexo masculino.
110.19 – HEMOCROMATOSE
1. Cobertura obrigatória para confirmação diagnóstica em pacientes nos quais as causas secundárias de sobrecarga de ferro tiverem sido excluídas e haja persistência de índice de saturação de transferrina maior que 45% em pelo menos duas dosagens.
Método de análise:
1. Detecção de mutações nos alelos C282Y e H63D do gene HFE por Reação em Cadeia da Polimerase (PCR) com ou sem polimorfismo do comprimento dos fragmentos de restrição (RFLP) ou PCR multiplex.
110.20 – HEMOFILIA A
1. Cobertura obrigatória para o aconselhamento genético, de pacientes do sexo masculino e com diagnóstico bioquímico de hemofilia no caso em que parentes de 1º e 2º graus do sexo feminino da linhagem materna tenham desejo de engravidar.
2. Cobertura obrigatória para o aconselhamento genético dos familiares do sexo feminino em risco (possibilidade de ser portadora assintomática – doença recessiva ligada ao X), apenas a partir do diagnóstico molecular do caso índice.
Método de análise utilizado de forma escalonada:
1. Nos casos em que a mutação genética já tenha sido identificada na família, realizar apenas a pesquisa da mutação específica.
2. No caso da forma grave de hemofilia, realizar:
a. PCR longa (Long-range PCR) ou PCR inversa (Inverse-shifting IS-PCR) para a detecção da inversão do íntron 22.
b. Sequenciamento Nova Geração dos 26 éxons do gene F8.
c. Nos casos em que o Sequenciamento Nova Geração não estiver disponível, realizar o Sequenciamento bidirecional por Sanger dos 26 éxons do gene F8.
3. No caso da forma leve ou moderada de hemofilia, realizar o Sequenciamento bidirecional por Sanger ou Sequenciamento de Nova Geração dos 26 éxons do gene F8.
110.21 – HEMOFILIA B
1. Cobertura obrigatória para o aconselhamento genético, de pacientes do sexo masculino e com diagnóstico bioquímico de hemofilia, no caso em que parentes de 1º e 2º graus do sexo feminino da linhagem materna tenham desejo de engravidar.
2. Cobertura obrigatória para o aconselhamento genético dos familiares do sexo feminino em risco (possibilidade de ser portadora assintomática de doença recessiva ligada ao X), apenas a partir do diagnóstico molecular do caso índice.
Método de análise utilizado de forma escalonada:
1. Nos casos em que a mutação genética já tenha sido identificada na família, realizar apenas a pesquisa da mutação específica.
2. Nos casos em que a mutação genética ainda não foi identificada na família, realizar Sequenciamento de Nova Geração ou Sequenciamento bidirecional por Sanger dos 8 éxons do gene F9.
110.22 – MUCOPOLISSACARIDOSE
1. Cobertura obrigatória para pacientes de ambos os sexos com diagnóstico enzimático de mucopolissacaridose I (alfa-L-iduronidase- gene IDUA) para aconselhamento genético de parentes de 1º e 2º graus com desejo de engravidar com finalidade de diagnóstico pré-natal.
2. Cobertura obrigatória para pacientes do sexo masculino com diagnóstico enzimático de mucopolissacaridose II (iduronato-2- sulfatase/gene IDS) para aconselhamento genético de parentes da linhagem materna de 1º, 2º e 3º graus com desejo de engravidar.
3. Cobertura obrigatória para aconselhamento genético de mulheres assintomáticas com história familiar de parentes de 1º, 2º e 3º graus do sexo masculino com mucopolissacaridose II e mutação patogênica identificada.
4. Cobertura obrigatória de feto de ambos os sexos em risco para mucopolissacaridose tipo I, quando a mutação do caso índice for conhecida.
Método de análise utilizado de forma escalonada:
1. No caso em que a mutação genética já tenha sido identificada na família, realizar apenas a pesquisa de mutação específica.
2. Para os casos do item 1 dos critérios de elegibilidade, realizar o Sequenciamento bidirecional por Sanger ou Sequenciamento de Nova Geração dos éxons do gene correspondente à mucopolissacaridose de acordo com análise enzimática identificada.
3. Para MPS II, caso o Sequenciamento bidirecional pelo método analítico de Sanger ou Sequenciamento de Nova Geração dos éxons do gene IDS não detecte alterações, realizar MLPA (Multiplex Ligation-dependent Probe Amplification) ou Hibridização Comparativa para pesquisa de deleções do cromossomo X.
4. Para mulheres em risco de serem portadoras de MPS II, com Sequenciamento bidirecional pelo método analítico de Sanger, Sequenciamento de Nova Geração e MLPA (Multiplex Ligation-dependent Probe Amplification) normais, realizar pesquisa de rearranjo entre o gene IDS e o pseudogene IDS2.
110.23 – NEOPLASIA ENDÓCRINA MÚLTIPLA TIPO I-MEN1
1. Cobertura obrigatória para pacientes de ambos os sexos, que apresentem pelo menos 2 (dois) critérios do Grupo I com ou sem história familiar de MEN1:
Grupo I:
a. Tumores das glândulas paratireoides;
b. Tumores da glândula pituitária;
c. Tumores endócrinos bem diferenciados do trato gastro-entero-pancreático.
2. Cobertura obrigatória para aconselhamento genético e teste da mutação familiar de pacientes assintomáticos, familiares de 1º, 2º ou 3º graus de caso índice com diagnóstico molecular de MEN1.
Método de análise utilizado de forma escalonada:
1. Nos casos em que a mutação genética já tenha sido identificada na família, realizar apenas a pesquisa da mutação específica.
2. Nos casos em que a mutação genética ainda não foi identificada na família, realizar Sequenciamento por Nova Geração do gene MEN1.
3. Se não for possível o item 2, realizar o Sequenciamento bidirecional por Sanger dos éxons do gene MEN1.
4. Para os casos onde os itens 2 ou 3 não forem conclusivos, realizar MLPA.
110.24 – NEOPLASIA ENDÓCRINA MÚLTIPLA TIPO 2A– MEN2A
1. Cobertura obrigatória para pacientes com diagnóstico de câncer medular de tireóide com ou sem história familiar.
2. Cobertura obrigatória para pacientes que preencham pelo menos um dos critérios do Grupo I e do Grupo II *:
Grupo I:
Pacientes com diagnóstico de:
a. Feocromocitoma;
b. Neuromas de mucosas;
c. Hiperparatireoidismo;
d. Hábito marfanóide.
Grupo II:
Parentes de 1º e 2º graus com diagnóstico de:
a. Carcinoma medular de tireóide;
b. Feocromocitoma;
c. Neuromas de mucosas;
d. Hiperparatireoidismo;
e. Hábito marfanóide.
*exceto em pacientes que apresentem apenas hábito marfanóide isoladamente nos Grupos I e II.
3. Cobertura obrigatória para o aconselhamento genético e teste da mutação familiar de familiares de 1º, 2º e 3º graus após o diagnóstico molecular do caso índice.
Método de análise utilizado de forma escalonada:
1. Nos casos em que a mutação genética já tenha sido identificada na família, realizar apenas a pesquisa da mutação específica.
2. Sequenciamento bidirecional pelo método analítico de Sanger dos éxons 5, 8, 10, 11, 13, 14, 15 e 16 do gene RET ou Sequenciamento de Nova Geração do gene RET.
OBS: Nos pacientes assintomáticos em que forem encontradas mutações patogênicas ou provavelmente patogênicas no gene RET a tireoidectomia profilática é de cobertura obrigatória, quando indicada pelo médico assistente.
110.25 – OSTEOGÊNESE IMPERFEITA
1. Cobertura obrigatória para pacientes sintomáticos com quadro clínico e radiológico sugestivo de alguma das formas de apresentação da doença com ou sem histórico familiar, com dosagem sérica de cálcio e fósforo normais e fosfatase alcalina normal ou aumentada quando os seus genitores ou o indivíduo sintomático tenham desejo de engravidar.
Método de análise utilizado de forma escalonada:
1. Pesquisa da mutação única c-14C-T por Sequenciamento bidirecional pelo método analítico de Sanger da regi o 5’UTR do gene IFITM5, a enas nos casos em que houver calcificação da membrana interóssea do antebraço ou perna, deslocamento da cabeça do rádio ou calo ósseo hiperplásico.
2. Realizar Sequenciamento de Nova Geração envolvendo os genes COL1A1, COL1A2, CRTAP, LEPR1 e PPIB.
3. No caso de não estar disponível o Sequenciamento de Nova Geração, realizar Sequenciamento por Sanger de maneira escalonada, conforme descrito abaixo:
I- Sequenciamento por Sanger do gene COL1A1.
II- Caso não seja encontrada alteração patogênica no item I, realizar Sequenciamento por Sanger do gene COL1A2.
110.26 – PAINEL DE GENES PARA CÂNCER DE MAMA E/OU OVÁRIO
1. Cobertura obrigatória para pacientes com diagnóstico atual ou prévio de câncer de ovário epitelial quando preencherem critérios para pelo menos 2 das seguintes síndromes: Síndrome de Câncer de Mama e Ovário Hereditários, Síndrome de Lynch e/ou Síndrome de Peutz-Jeghers.
2. Cobertura obrigatória para pacientes com diagnóstico atual ou prévio de câncer de mama quando preencherem critérios para pelo menos 2 das seguintes síndromes: Síndrome de Câncer de Mama e Ovário Hereditários, Síndrome de Cowden, Síndrome de Li-Fraumeni, Síndrome de Câncer Gástrico Difuso Hereditário e/ou Síndrome de Peutz-Jeghers.
3. Cobertura obrigatória para pacientes com diagnóstico atual ou prévio de câncer de mama e/ou ovário epitelial com critérios para câncer de mama e ovário hereditários e que tenham resultado negativo na análise de mutações germinativas de BRCA1 e BRCA2 por sequenciamento e MLPA.
Método de análise:
1. Análise de DNA genômico com painel NGS incluindo os genes: ATM, BRCA1, BRCA2, BRIP1, CDH1, CHEK2, MLH1, MSH2, MSH6, PALB2, PMS2, PTEN, RAD51C, RAD51D, STK11, TP53.
2. Em caso de resultado negativo, se o painel NGS não estiver validado para análise de CNVs, realizar análise com MLPA dos genes: BRCA1, BRCA2, CDH1, EPCAM, MLH1, MSH2, MSH6, PALB2, STK11.
OBS1. Quando forem encontradas mutações patogênicas ou provavelmente patogênicas nos genes BRCA1, BRCA2, CDH1, PALB2, PTEN, TP53, mesmo em pacientes assintomáticas, a mastectomia redutora de risco, bem como a reconstrução das mamas são de cobertura obrigatória da mesma forma que a cobertura prevista para pacientes com diagnóstico de câncer, quando indicado pelo médico assistente.
OBS2. Quando forem encontradas mutações patogênicas ou provavelmente patogênicas nos genes BRCA1, BRCA2, BRIP1, MLH1, MSH2, MSH6, PMS2, RAD51C,
RAD51D, STK11, mesmo em pacientes assintomáticas, a salpingo-ooforectomia redutora de risco, é de cobertura obrigatória da mesma forma que a cobertura prevista para pacientes com diagnóstico de câncer, quando indicado pelo médico assistente.
OBS3. Quando forem encontradas mutações patogênicas ou provavelmente patogênicas nos genes ATM, BRCA1, BRCA2, CDH1, PALB2, PTEN, TP53, e que não tenham sido submetidas a mastectomia redutora de risco, a realização de ressonância magnética periódica é de cobertura obrigatória, quando indicado pelo médico assistente.
110.27 – POLIPOSE COLÔNICA
1. Cobertura obrigatória para pacientes com a forma clássica (mais de 100 pólipos) ou atenuada (10 a 100 pólipos) de polipose colônica.
2. Cobertura obrigatória para o aconselhamento genético e teste da mutação familiar de familiares de 1º, 2º e 3º graus após o diagnóstico molecular de mutação patogênica no gene APC no caso índice.
3. Cobertura obrigatória para o aconselhamento genético de irmãos e irmãs de pacientes que já tenham mutação patogênica identificada no gene MUTYH.
Método de análise utilizado de forma escalonada:
1. Nos casos em que a mutação já tenha sido identificada na família, realizar apenas a pesquisa da mutação específica.
2. Nos casos não enquadrados no item acima, realizar o Sequenciamento de Nova Geração dos éxons dos genes APC e MUTYH.
3. Nos casos em que o diagnóstico não for estabelecido através do item anterior, realizar MLPA (Multiplex Ligation dependent Probe Amplification) do gene APC.
OBS 1: Nos pacientes com polipose adenomatosa profusa (>100 pólipos) ou pólipos não rastreáveis por colonoscopia e naqueles com mutação patogênica ou provavelmente patogênica identificada em APC, a colectomia total é de cobertura obrigatória, quando indicada pelo médico assistente.
110.28 – SÍNDROME CHARGE
1. Cobertura obrigatória pacientes de ambos os sexos com cariótipo normal e com pelo menos 2 características maiores e pelo menos 2 características menores da síndrome CHARGE.
Características maiores:
a. Microftalmia ou coloboma ocular (coloboma de íris e/ou retina e/ou coróide e/ou disco);
b. Atresia ou estenose de coana (uni ou bilateral);
c. Disfunção de nervo craniano (hiposmia e/ou anosmia e/ou paralisia facial e/ou hipoplasia do nervo auditivo e/ou dificuldade de deglutição com aspiração);
d. Alterações de orelha característica de charge (orelha displásica, malformações ossiculares em orelha média, malformação de mondini, anormalidades do osso temporal, ausência ou hipoplasia de canais semicirculares).
Características menores:
a. Hipoplasia genital ou hipogonadismo hipogonadotrófico;
b. Atraso do desenvolvimento neuropsicomotor;
c. Malformação cardiovascular;
d. Déficit de crescimento;
e. Fenda orofacial;
f. Fístula traqueoesofágica;
g. Dismorfismos faciais.
Método de análise utilizado de forma escalonada:
1. Excluir anomalias cromossômicas no cariótipo.
2. Pesquisa de mutação no gene CHD7 por Sequenciamento bidirecional por Sanger ou Sequenciamento de Nova Geração.
110.29 – SÍNDROME DE ANGELMAN E SÍNDROME DE PRADER-WILLI
1. Cobertura obrigatória para pacientes de ambos os sexos com atraso do desenvolvimento e manifestações clínicas sugestivas da doença (fenótipo) de Síndrome de Angelman ou Síndrome de Prader-Willi.
2. Cobertura obrigatória para o aconselhamento genético de familiar de 1º grau assintomático do caso índice com diagnóstico molecular de mutação no gene UBE3A (para Síndrome de Angelman).
Método de análise utilizado de forma escalonada:
1. Nos casos de Síndrome de Angelman em que a mutação genética já tenha sido identificada na família, realizar apenas a pesquisa da mutação específica no gene UBE3A.
2. Para confirmação diagnóstica em pacientes sintomáticos com suspeita de Síndrome de Angelman ou Síndrome de Prader-Willi, realizar teste de metilação da região cromossômica do gene SNRPN (15q11.2):
a. Se metilação alterada, realizar FISH (Hibridação In Situ Fluorescente) ou MLPA (Multiplex Ligation-dependent Probe Amplification) para pesquisa de deleção da região 15q11.2
b. Se FISH (Hibridação In Situ Fluorescente) ou MLPA (Multiplex Ligation-dependent Probe Amplification) forem normais, realizar Análise de Microssatélites para pesquisa de dissomia uniparental da região 15q11.2.
3. Para confirmação diagnóstica em pacientes sintomáticos com suspeita de Síndrome de Angelman e teste de metilação normal, realizar a pesquisa de mutações nos éxons do UBE3A por Sequenciamento bidirecional pelo método analítico de Sanger ou Sequenciamento de Nova Geração dos éxons do gene UBE3A.
110.30 – SINDROME DE COWDEN
1. Cobertura obrigatória para pacientes de ambos os sexos com macrocefalia e pelo menos um dos seguintes critérios:
a. Pelo menos um câncer do espectro da Síndrome de Cowden (câncer de mama, câncer de endométrio, câncer de tireoide folicular);
b. Pelo menos uma lesão benigna típica da Síndrome de Cowden entendida como presença de pelo menos um dos itens abaixo:
i. múltiplos hamartomas gastrointestinais;
ii. ganglioneuromas;
iii. pigmentação macular da glande do pênis;
iv. um triquilemoma comprovado por biópsia;
v. múltiplas queratoses palmo-plantares;
vi. papilomatose multifocal ou extensa de mucosa oral;
vii. inúmeras pápulas faciais.
c. Com diagnóstico de transtorno do espectro autista.
2. Cobertura obrigatória para pacientes de ambos os sexos sem macrocefalia com diagnóstico atual ou prévio de pelo menos três lesões benignas ou malignas da Síndrome de Cowden entendida como presença de pelo menos três dos itens abaixo:
a. Câncer de mama;
b. Câncer de endométrio;
c. Câncer de tireoide folicular;
d. Múltiplos hamartomas gastrointestinais;
e. Ganglioneuromas;
f. Pigmentação macular da glande do pênis;
g. Triquilemoma comprovado por biópsia;
h. Múltiplas queratoses palmo-plantares;
i. Papilomatose multifocal ou extensa de mucosa oral;
j. Inúmeras pápulas faciais.
3. Cobertura obrigatória para pacientes de ambos os sexos com diagnóstico atual ou prévio de pelo menos quatro dos critérios menores da Síndrome de Cowden
(Carcinoma de células renais, Câncer de cólon, Câncer de tireóide a ilífero, ≥3 Acantoses esofágicas glicogênicas, lipomas, lipomatose testicular, adenoma de tireoide, nódulo de tireoide ou bócio multinodular de tireoide, anomalias vasculares incluindo múltiplas anomalias venosas intracranianas de desenvolvimento, Déficit cognitivo com QI ≤75, transtorno do espectro autista).
4. Cobertura obrigatória para pacientes de ambos os sexos com um câncer do espectro da Síndrome de Cowden (câncer de mama, câncer de endométrio, câncer de tireoide folicular) e mais três das lesões menores da Síndrome de Cowden (carcinoma de células renais, câncer de cólon, câncer de tireóide papilífero, ≥3 Acantoses esofágicas glicogênicas, lipomas, lipomatose testicular, adenoma de tireoide, nódulo de tireoide ou bócio multinodular de tireoide, anomalias vasculares incluindo múltiplas anomalias venosas intracranianas de desenvolvimento, Déficit cognitivo com QI ≤ 75, transtorno do espectro autista).
5. Cobertura obrigatória para pacientes de ambos os sexos com diagnóstico atual ou prévio de uma lesão benigna típica da Síndrome de Cowden (múltiplos hamartomas gastrointestinais, ganglioneuromas, pigmentação macular da glande do pênis, triquilemomas, múltiplas queratoses palmo-plantares, papilomatose de mucosa oral, inúmeras pápulas faciais) e mais três das lesões menores da Síndrome de Cowden (Carcinoma de células renais, Câncer de cólon, Câncer de tireoide a ilífero, ≥ 3 Acantoses esof gicas glicogênicas, li omas, li omatose testicular, adenoma de tireoide, nódulo de tireoide ou bócio multinodular de tireoide, anomalias vasculares incluindo múltiplas anomalias venosas intracranianas de desen ol imento, Déficit cogniti o com QI ≤ 75, transtorno do es ectro autista).
6. Cobertura obrigatória para pacientes de ambos os sexos com diagnóstico de Bannayan-Riley-Ruvalcaba ou doença de Lhermitte-Duclos no adulto.
7. Cobertura obrigatória para pacientes de ambos os sexos sem macrocefalia com diagnóstico atual ou prévio de pelo menos dois triquilemomas comprovados por biópsia.
8. Cobertura obrigatória para indivíduos maiores de 18 anos, diagnosticados ou não com câncer, com ou sem achados clínicos da Síndrome de Cowden,
independente do sexo, quando houver mutação deletéria em PTEN em familiar de 1º, 2º ou 3º graus.
Método de análise utilizado de forma escalonada:
1. Sequenciamento bidirecional pelo método de Sanger ou Sequenciamento de Nova Geração dos éxons do gene PTEN.
2. Se o item anterior for negativo, realizar MLPA.
3. Se os itens anteriores forem negativos, investigar mutações na região promotora do gene por Sequenciamento bidirecional pelo método de Sanger ou outro método..
OBS 1: Nas pacientes em que forem encontradas mutações patogênicas ou provavelmente patogênicas no gene PTEN, mesmo que assintomáticas, a mastectomia e a histerectomia redutoras de risco, bem como a reconstrução das mamas são de cobertura obrigatória da mesma forma que a cobertura prevista para pacientes com diagnóstico de câncer, quando indicado pelo médico assistente. Caso a beneficiária não deseje realizar mastectomia a ressonância magnética das mamas anual é de cobertura obrigatória.
110.31 – SÍNDROME DE HIPOFOSFATASIA
1. Cobertura obrigatória para o aconselhamento genético de pacientes sintomáticos com quadro clínico e radiológico compatível com alguma das formas de apresentação da doença com ou sem histórico familiar, com dosagem sérica de fosfatase alcalina diminuída, quando os seus genitores ou o indivíduo sintomático desejarem uma gestação.
Método analítico:
1. Sequenciamento bidirecional por Sanger ou Sequenciamento de Nova Geração dos éxons do gene TNSAP.
110.32 – SÍNDROME DE LI-FRAUMENI
1. Cobertura obrigatória para pacientes de ambos os sexos com diagnóstico atual ou prévio de sarcoma antes dos 45 anos e história familiar de câncer em um familiar de 1º grau antes dos 45 anos e mais um caso de câncer em um familiar de 1º ou 2º graus do mesmo lado da família com câncer antes dos 45 anos ou sarcoma em qualquer idade.
2. Cobertura obrigatória para pacientes com diagnóstico atual ou prévio de tumor característico da Síndrome de Li-Fraumeni (Sarcoma de partes moles, osteossarcoma, tumor de Sistema Nervoso Central, câncer de mama, carcinoma adrenocortical, leucemia, adenocarcinoma de pulmão) antes dos 46 anos quando preenchido um dos seguintes critérios:
a. Diagnóstico de outro tumor primário no mesmo indivíduo típico da Síndrome de Li-Fraumeni (Sarcoma de partes moles, osteossarcoma, tumor de SNC, câncer de mama, carcinoma adrenocortical, leucemia, adenocarcinoma de pulmão);
b. Um familiar de 1º ou 2º graus com câncer antes dos 56 anos;
c. Um familiar de 1º ou 2º graus com múltiplos tumores primários característicos da Síndrome de Li-Fraumeni (Sarcoma de partes moles, osteossarcoma, tumor de Sistema Nervoso Central, câncer de mama, carcinoma adrenocortical, leucemia, adenocarcinoma de pulmão). Se o caso índice tiver sido
diagnosticado com câncer de mama, o familiar deverá ter desenvolvido outro tumor do espectro Li-fraumeni diferente de câncer de mama.
3. Cobertura obrigatória para pacientes de ambos os sexos com diagnóstico atual ou prévio de carcinoma adrenocortical em qualquer idade e independente da história familiar de câncer.
4. Cobertura obrigatória para pacientes de ambos os sexos com diagnóstico atual ou prévio de carcinoma de plexo coróide em qualquer idade e independente da história familiar de câncer.
5. Cobertura obrigatória para pacientes de ambos os sexos com diagnóstico atual ou prévio de rabdomiosarcoma anáplasico em qualquer idade e independente da história familiar de câncer.
6. obertura obrigatória ara acientes com diagnóstico de câncer de mama ≤ 35 anos de idade.
7. Cobertura obrigatória para o aconselhamento genético e teste da mutação familiar dos familiares de 1º, 2º ou 3º graus assintomáticos quando o diagnóstico molecular de Síndrome de Li-Fraumeni tiver sido confirmado na família.
Método de análise utilizado de forma escalonada:
Para pacientes enquadrados nos itens 1, 2, 3, 4 e 5:
1. Análise da mutação específica p.Arg337His no gene TP53.
2. Se o item anterior for negativo, realizar Sequenciamento bidirecional por Sanger ou Sequenciamento de Nova Geração dos éxons do gene TP53.
3. Se o item anterior for negativo, realizar pesquisa de rearranjos por MLPA.
Para pacientes enquadrados no item 6:
1. Realizar inicialmente a pesquisa de mutação dos genes BRCA1 e BRCA2 conforme método escalonado descrito na diretriz específica de BRCA1 e BRCA2.
2. Não sendo identificadas mutações nos genes BRCA1 e/ou BRCA2, realizar a mutação específica Arg337His no gene TP53.
3. Se a análise da mutação p.Arg337His for negativa, realizar Sequenciamento bidirecional por Sanger ou Sequenciamento de Nova Geração dos éxons do gene TP53.
4. Se o item anterior for negativo, realizar pesquisa de rearranjos por MLPA.
Para pacientes enquadrados no item 7:
1. Nos casos de Síndrome de Li-Fraumeni em que a mutação genética já tenha sido identificada na família, realizar apenas a pesquisa da mutação específica já identificada.
OBS 1: Nos pacientes em que forem encontradas mutações patogênicas ou provavelmente patogênicas do domínio de ligação do gene TP53 (mutações clássicas), mesmo que assintomáticos, a mastectomia redutora de risco, bem como a reconstrução das mamas são de cobertura obrigatória da mesma forma que a cobertura prevista para pacientes com diagnóstico de câncer, quando indicado pelo médico assistente. Caso a beneficiária não deseje realizar mastectomia a ressonância magnética das mamas anual é de cobertura obrigatória. Para portadoras da mutação p.Arg337His de TP53 a ressonância magnética das mamas anual é de cobertura obrigatória.
110.33 – SÍNDROME DE LYNCH – CÂNCER COLORRETAL NÃO POLIPOSO HEREDITÁRIO (HNPCC)
1. Cobertura obrigatória para pacientes com câncer colorretal que preencham um dos Critérios de Bethesda listados abaixo:
a. Paciente diagnosticado com câncer colorretal com menos de 50 anos;
b. Presença de pelo menos 2 tumores colorretais sincrônicos, metacrônicos ou pelo menos 2 outras neoplasias extracolônicas associadas à Síndrome de Lynch (HNPCC) diagnosticadas em qualquer idade;
c. Paciente diagnosticado com câncer colorretal com instabilidade de microssatélites de alto grau (MSI-H) diagnosticado com menos de 60 anos;
d. Paciente diagnosticado com câncer colorretal com um ou mais parentes de 1º grau acometidos por neoplasias associadas à Síndrome de Lynch (HNPCC), sendo uma destas diagnosticada antes dos 50 anos;
e. Paciente diagnosticado com câncer colorretal com dois ou mais parentes de 1º grau acometidos por neoplasias associadas à Síndrome de Lynch (HNPCC) independentemente da idade.
2. Cobertura obrigatória para pacientes com diagnóstico de tumores do espectro da Síndrome de Lynch (adenocarcinoma colorretal, adenocarcinoma de endométrio, carcinoma urotelial [ureter e de pelve-renal], adenocarcinoma de ovário, adenocarcinoma gástrico; câncer de intestino delgado; glioblastoma; adenocarcinoma sebáceo; câncer do trato biliar e câncer de pâncreas) desde que preenchidos todos os critérios de Amsterdam II para a história familiar.
Critérios de Amsterdam II:
a. Três membros do mesmo lado da família, dois dos quais sejam parentes de 1º grau, com câncer do espectro da S. Lynch (conforme descrito acima);
b. Duas gerações sucessivas acometidas;
c. Um desses familiares com câncer diagnosticado com menos de 50 anos;
d. Excluído o diagnóstico de polipose adenomatosa familiar.
3. Cobertura obrigatória para mulheres com adenocarcinoma de endométrio diagnosticado com 50 anos ou menos, mesmo que isoladamente e independente de história familiar.
4. Cobertura obrigatória para o aconselhamento genético e teste da mutação familiar de familiares de 1º, 2º e 3º graus após o diagnóstico molecular de mutação patogênica ou provavelmente patogênica no caso índice.
Método de análise utilizado de forma escalonada:
1. Nos casos em que a mutação já foi identificada na família, realizar apenas a pesquisa da mutação específica.
2. No caso de pacientes enquadrados nos critérios 1 e 3:
2.1. Realizar Imunohistoquímica (IHQ) para MLH1, MSH2, MSH6 e PMS2;
I. No caso de imunohistoquímica proficiente (todos os quatro marcadores presentes no tumor), realizar instabilidade de microssatélites.
a. Se não houver instabilidade de microssatélite em nenhum dos cinco marcadores encerra-se a investigação.
b. Se instabilidade alta (2 em 5 marcadores instáveis), realizar sequenciamento de nova geração NGS para MLH1, MSH2, MSH6 e PMS2*.
c. Se o item anterior for negativo realizar MLPA para pesquisa de rearranjos MLH1 e MSH2.
d. Se o item anterior for negativo realizar MLPA para pesquisa de rearranjos MSH6 e EPCAM.
II. Se houver perda de expressão da proteína MLH1 ou MLH1+PMS2 na IHQ, realizar pesquisa da mutação V600E do gene BRAF ou metilação do promotor do gene MLH1 no tumor para diferenciar instabilidade de origem somática ou hereditária:
a. Na ausência de mutação V600E do gene BRAF ou na ausência de metilação do promotor do gene MLH1, realizar sequenciamento de nova geração NGS para MLH1, MSH2, MSH6 e PMS2*.
b. Se o item anterior for negativo realizar MLPA para pesquisa de rearranjos MLH1 e MSH2.
c. Se o item anterior for negativo realizar MLPA para pesquisa de rearranjos MSH6 e EPCAM.
III. Se houver perda de expressão da proteína MSH2 ou MSH6 ou PMS2 ou dos quatro marcadores na IHQ, realizar Sequenciamento de Nova Geração para MLH1, MSH2, MSH6 e PMS2.
a. Se o item anterior for negativo realizar MLPA para pesquisa de rearranjos MLH1 e MSH2.
b. Se o item anterior for negativo realizar MLPA para pesquisa de rearranjos MSH6 e EPCAM.
c. Se resultado da investigação do item anterior for negativo, realizar MLPA para MLH1.
3. No caso de pacientes enquadrados no critério 2:
a. Realizar Sequenciamento de Nova Geração envolvendo os genes MLH1, MSH2, MSH6, PMS2 e EPCAM.
b. Se o item anterior for negativo realizar MLPA para pesquisa de rearranjos MLH1 e MSH2.
c. Se o item anterior for negativo realizar MLPA para pesquisa de rearranjos MSH6 e EPCAM.
OBS 1: Nos pacientes em que forem encontradas mutações patogênicas ou provavelmente patogênicas nos genes acima, mesmo que assintomáticos, a histerectomia e a salpigo-ooforectomia redutoras de risco, devem ser discutidas entre a paciente e seu médico assistente e caso optem pelas cirurgias estas devem ser de cobertura obrigatória da mesma forma que a cobertura prevista para pacientes com diagnóstico de câncer. Pacientes com mutação patogênica nos genes MLH1 ou MSH2 ou EPCAM devem realizar colonoscopia a cada 1 ou 2 anos a partir dos 20 a 25 anos ou 2 a 5 anos antes do caso mais jovem identificado na família. Pacientes com mutação patogênica nos genes MSH6 ou PMS2 devem realizar colonoscopia a cada 1 ou 2 anos a partir dos 25 a 30 anos ou 2 a 5 anos antes do caso mais jovem identificado na família.
(*) OBS 2: O gene PMS2 apresenta pseudogenes de alta similaridade, dificultando o estudo genético. O uso das técnicas usuais pode levar a falsos-positivos e falsos-negativos. O método utilizado para estudo desse gene deve ser o PCR de longa distância (long-range PCR), seguido de sequenciamento por NGS ou Sanger.
110.34 – SÍNDROME DE MARFAN
1. Cobertura obrigatória para pacientes de ambos os sexos com escore sistêmico ≤ 7 quando reenchido a enas um dos critérios abaixo (caso o paciente preencha ambos os critérios abaixo, a cobertura do diagnóstico molecular não é obrigatória):
a. Ectopia Lentis: Subluxação ou Luxação de cristalino;
b. Dilataç o da rai da aorta com ecocardiograma que demonstre escore Z ≥ 2 em pacientes acima de 20 anos ou Z ≥ 3 em acientes abaixo de 20 anos.
Cálculo do Escore Sistêmico:
i. Sinal do punho e do polegar – 3 (punho ou polegar – 1)
ii. Peito carenado – 2 (peito escavado ou assimetria de tórax – 1)
iii. Deformidade dos pés – 2 (pés planos – 1)
iv. Pneumotórax – 2
v. Ectasia dural – 2
vi. Protrusão acetabular – 2
vii. Relação Segmento Superior/Segmento Inferior reduzida e Relação Envergadura/Estatura aumentada e escoliose leve – 1
viii. Escoliose ou cifose toracolombar – 1
ix. Extensão reduzida do cotovelo – 1
x. Características faciais (3/5) – 1 (dolicocefalia, enoftalmia, fendas palpebrais com inclinação para baixo, hipoplasia malar, retrognatia)
xi. Estrias na pele – 1
xii. Miopia > 3 dpt – 1
xiii. Prolapso de valva mitral (todos os tipos) – 1
Total = 20 ontos; escore ≥ 7 indica en ol imento sistêmico; SS/SI = razão do segmento superior/segmento inferior.
2. Cobertura obrigatória para indivíduos assintomáticos em risco de herdarem a mutação e de desenvolverem a Síndrome Marfan que apresentem familiares de 1º, 2º ou 3º graus confirmados por análise molecular.
Método de análise utilizado de forma escalonada:
1. Nos casos em que a mutação genética já tenha sido identificada na família, realizar apenas a pesquisa da mutação específica.
2. Pesquisa de mutação no gene FBN1 por Sequenciamento de Nova Geração
3. Se não for possível realizar o Sequenciamento de Nova Geração, realizar o Sequenciamento bidirecional por Sanger
4. Nos casos em que o diagnóstico não for estabelecido através do item anterior, realizar MLPA (Multiplex Ligation-dependent Probe Amplification) para o gene FBN1.
110.35 – SÍNDROME DE NOONAN
1. Cobertura obrigatória para pacientes do sexo feminino com ou sem histórico familiar da doença, quando o paciente apresentar manifestações clínicas sugestivas da doença e excluída a Síndrome de Turner.
2. Cobertura obrigatória para pacientes do sexo masculino com ou sem histórico familiar da doença, quando o paciente apresentar manifestações clínicas sugestivas da doença.
Método de análise utilizado de forma escalonada:
1. Realizar Sequenciamento de Nova Geração envolvendo os genes PTPN1, SOS1, RAF1, RIT1 e KRAS.
2. No caso de não estar disponível o Sequenciamento de Nova Geração, realizar Sequenciamento por Sanger de maneira escalonada, conforme descrito abaixo:
a. Sequenciamento bidirecional por Sanger dos éxons do gene PTPN11.
b. Sequenciamento bidirecional por Sanger dos éxons do gene SOS1.
c. Sequenciamento bidirecional por Sanger dos éxons do gene RAF1.
d. Sequenciamento bidirecional por Sanger dos éxons do gene RIT1.
e. Sequenciamento bidirecional por Sanger dos éxons do gene KRAS.
110.36 – SÍNDROME DE RETT
1. Cobertura obrigatória para pacientes do sexo feminino e que apresentem inicialmente um período de desenvolvimento normal e um período de regressão do desenvolvimento neuropsicomotor seguido por recuperação parcial ou estabilização e que se enquadrem em um dos itens abaixo:
a. Para as pacientes com Síndrome de Rett Clássica que preencham todos os critérios do Grupo I e nenhum dos critérios do Grupo II;
b. Para as pacientes com Síndrome de Rett Atípica que preencham pelo menos 2 critérios do Grupo I e 5 do Grupo III.
Grupo I (Critérios principais):
a. Perda total ou parcial de habilidades manuais intencionais adquiridas ao longo do desenvolvimento;
b. Perda total ou parcial de fala ou habilidades de comunicação adquiridas ao longo do desenvolvimento, como a lalação;
c. Alterações de marcha: dispraxia ou ausência da habilidade;
d. Movimentos estereotipados de mão.
Grupo II (Critérios de exclusão):
a. Diagnóstico prévio de lesão cerebral secundária a trauma perinatal ou pós-natal, doenças neurometabólicas ou infecções com sequelas neurológicas;
b. Desenvolvimento neuropsicomotor com atraso importante nos primeiros 6 meses de vida, sem aquisição de marcos de desenvolvimento.
Grupo III (Critérios de apoio):
a. Distúrbios respiratórios (apnéia e/ou hiperpnéia) quando a paciente encontra-se acordada;
b. Bruxismo quando a paciente encontra-se acordada;
c. Distúrbios de padrão de sono;
d. Tônus muscular alterado;
e. Distúrbios vasomotores periféricos;
f. Cifose e/ou escoliose;
g. Déficit de crescimento;
h. Mãos e pés pequenos e frios;
i. Risos ou gritos sem motivação aparente;
j. Pouca resposta a estímulos dolorosos;
k. Comunicação intensa com o olhar.
Método de análise utilizado de forma escalonada:
1. Realizar Sequenciamento de Nova Geração ou Sanger de toda região codificante do gene MECP2;
2. Caso não tenha sido identificada mutação patogênica no item anterior, realizar análise de deleções e duplicações no gene MECP2 por MLPA.
110.37 – SÍNDROME DE WILLIAMS-BEUREN
1. Cobertura obrigatória para pacientes com suspeita de Williams-Beuren (del7q11) que apresentem manifestações clínicas sugestivas da doença (fenótipo).
Método de análise utilizado de forma escalonada:
1. Preferencialmente por MLPA (Multiplex Ligation-dependent Probe Amplification), ou FISH (Hibridação In Situ Fluorescente) quando o MLPA (Multiplex Ligation-dependent Probe Amplification) não estiver disponível.
2. No caso em que o diagnóstico não tenha sido confirmado através da Hibridação in situ fluorescente (FISH), realizar MLPA (Multiplex Ligation-dependent Probe Amplification).
110.38 – SÍNDROME DO CÂNCER GÁSTRICO DIFUSO HEREDITÁRIO
1. Cobertura obrigatória para indivíduos de ambos os sexos com diagnóstico de câncer gástrico difuso e com pelo menos um familiar de 1º, 2º ou 3º graus com câncer gástrico difuso, sendo um deles com diagnóstico em idade ≤ 50 anos.
2. Cobertura obrigatória para indivíduos de ambos os sexos com diagnóstico de câncer gástrico difuso com pelo menos dois familiares de 1º ou 2º graus com câncer gástrico difuso em qualquer idade.
3. Cobertura obrigatória para indivíduos de ambos os sexos com diagnóstico de câncer g strico difuso em idade ≤ 40 anos.
4. Cobertura obrigatória para indivíduos de ambos os sexos com diagnóstico de câncer gástrico difuso e um caso de carcinoma de mama do tipo lobular sendo pelo menos um deles diagnosticado em idade ≤ 50 anos.
5. Cobertura obrigatória para o aconselhamento genético e teste da mutação familiar dos familiares de 1º, 2º ou 3º graus assintomáticos quando o diagnóstico molecular de Síndrome do Câncer Gástrico Difuso Hereditário tiver sido confirmado na família.
Método de análise utilizado de forma escalonada:
1. Nos casos em que a mutação já foi identificada na família, realizar apenas a pesquisa da mutação específica.
2. Sequenciamento de Nova Geração dos éxons do gene CDH1.
3. Nos casos em que o Sequenciamento de Nova Geração não estiver disponível, realizar o Sequenciamento bidirecional por Sanger do gene CDH1.
4. Nos casos em que o diagnóstico não for estabelecido através dos itens anteriores, realizar MLPA (Multiple Ligation Dependent Probe Amplification) do gene CDH1.
OBS 1: Nos pacientes em que forem encontradas mutações patogênicas ou provavelmente patogênicas no gene CDH1, mesmo que assintomáticos, a mastectomia redutora de risco e gastrectomia total, bem como a reconstrução das mamas são de cobertura obrigatória da mesma forma que a cobertura prevista para pacientes com diagnóstico de câncer, quando indicado pelo médico assistente. Caso a beneficiária não deseje realizar mastectomia a ressonância magnética das mamas anual é de cobertura obrigatória.
110.39 – SÍNDROMES DE ANOMALIAS CROMOSSÔMICAS SUBMICROSCÓPICAS NÃO RECONHECÍVEIS CLINICAMENTE (ARRAY)
1. Cobertura obrigatória para pacientes de ambos os sexos com cariótipo normal e suspeita clínica de anomalias cromossômicas submicroscópicas quando preenchidos pelo menos dois dos seguintes critérios:
a. Deficiência intelectual ou atraso neuropsicomotor;
b. Presença de pelo menos uma anomalia congênita maior ou pelo menos três menores;
c. Baixa estatura ou déficit pondero-estatural.
2. Cobertura obrigatória para pacientes de ambos os sexos com cariótipo alterado quando preenchidos um dos seguintes critérios:
a. Cromossomo marcador;
b. Translocações ou inversões cromossômicas aparentemente balanceadas identificadas pelo cariótipo com fenótipo anormal;
c. Presença de material cromossômico adicional de origem indeterminada;
d. Presença de alteração cromossômica estrutural (para determinar tamanho e auxiliar na correlação genótipo-fenótipo).
3. Cobertura obrigatória para aconselhamento genético dos pais em que tenha sido identificada uma variante de significado incerto no CGH-Array (Hibridização Genômica Comparativa) ou SNP-array (Polimorfismo de um único nucleotídeo) no caso índice.
4. Cobertura obrigatória para aconselhamento genético dos pais em que tenha sido identificada uma variação no CGH-Array (Hibridização Genômica Comparativa) por provável micro-rearranjo (translocação equilibrada ou inversões) no caso índice.
Método de análise utilizado de forma escalonada:
Nos pacientes enquadrados nos itens 1 e 2 e 3:
1. Realizar CGH- Array (Hibridização Genômica Comparativa) ou SNP-array (Polimorfismo de um único nucleotídeo) do caso índice.
2. Em caso de se identificar uma variante de significado incerto, a cobertura será obrigatória de CGH- Array (Hibridização Genômica Comparativa) ou SNP-array (Polimorfismo de um único nucleotídeo) dos pais do caso índice.
3. Em caso de resultado negativo, realizar o Sequenciamento Completo do Exoma.
Nos pacientes (pais do caso índice) enquadrados no item 4:
1. Realizar cariótipo.
2. Nos casos em que o diagnóstico não for confirmado através do item anterior, realizar FISH (Hibridação In Situ Fluorescente).
110.40 – SÍNDROMES DE DELEÇÕES SUBMICROSCÓPICAS RECONHECÍVEIS CLINICAMENTE
1. Cobertura obrigatória para pacientes de ambos os sexos com suspeita clínica de Wolf-Hirschhorn (del4p) ou Cri du Chat (del5p) ou Deleção 1p36 ou Smith-Magenis (del17p11) ou Deleção 22q11 ou Miller-Dieker (del17p13) ou WAGR(del11p13), quando preenchidos todos os seguintes critérios:
a. apresente cariótipo normal;
b. manifestações clínicas sugestivas da doença (fenótipo).
2. Cobertura obrigatória para o aconselhamento genético de familiar com cariótipo normal e que possuam parentes de 1o e 2o graus com diagnóstico molecular ou citogenético (Cariótipo ou FISH – Hibridação In Situ Fluorescente) de Wolf-Hirschhorn (del4p) ou Cri du Chat (del5p) ou Deleção 1p36 ou Smith-Magenis (del17p11) ou Deleção 22q11 ou Miller-Dieker (del17p13) ou WAGR (del11p13).
Método de análise utilizado de forma escalonada:
1. A tecnologia utilizada para o teste deve ser projetada para detectar a deleção da região crítica para a doença por FISH (Hibridação In Situ Fluorescente) ou MLPA (Multiplex Ligation-dependent Probe Amplification).
2. Nos casos em que o diagnóstico não tenha sido confirmado através dos métodos analíticos anteriores, realizar CGH- Array (Hibridização Genômica Comparativa) ou SNP-array (Polimorfismo de um único nucleotídeo).
110.41 – TRANSTORNO DO ESPECTRO AUTISTA
1. Cobertura obrigatória para pacientes de ambos os sexos com manifestações clínicas sugestivas de Transtorno do Espectro Autista, quando presentes pelo menos um dos critérios do Grupo I e nenhum dos critérios do Grupo II:
Grupo I:
a. Deficiência intelectual;
b. Crises convulsivas;
c. Malformação do Sistema Nervoso Central;
d. Dismorfias;
e. Microcefalia ou macrocefalia.
Grupo II:
a. Autismo isolado;
b. Alterações identificadas no cariótipo;
c. Síndrome do X-Frágil.
Método de análise utilizado de forma escalonada:
1. Excluir anomalias cromossômicas no cariótipo e Síndrome do X Frágil
2. Se não forem encontradas alterações no item anterior realizar CGH- Array (Hibridização Genômica Comparativa) ou SNP-array do caso índice.
3. Em caso de se identificar uma variante de significado incerto, a cobertura será obrigatória de CGH-Array (Hibridização Genômica Comparativa) ou SNP-array dos pais do caso índice.
110.42 – SÍNDROME DE PEUTZ-JEGHERS
1. Cobertura obrigatória da análise genética para pacientes com pelo menos duas das seguintes características:
a. lesões peri-orais, orais, genitais e/ou digitais hiperpigmentadas
b. 2 ou mais pólipos gastro-intestinais hamartomatosos de Peutz-Jeghers confirmados por histologia
c. familiar de 1º, 2º, ou 3º grau com diagnóstico clínico de Síndrome de Peutz-Jeghers.
2. Cobertura obrigatória para familiares de 1º, 2º, ou 3º grau de um indivíduo com mutação germinativa patogênica identificada no gene STK11.
Método de análise utilizado de forma escalonada:
1. Nos casos em que a mutação genética já foi identificada na família, realizar apenas a pesquisa da mutação específica.
2. Nos casos de pacientes com suspeita clínica ou diagnóstico clínico conforme critérios acima:
a. Sequenciamento de Nova Geração (NGS) de toda região codificadora e das junções intron-exon do gene STK11 e se NGS não estiver disponível realizar sequenciamento bidirecional de toda região codificadora e das junções intron-exon por Sanger;
b. Em caso de resultado negativo no item 3.2.a, realizar MLPA do gene STK11. Deleções de um éxon simples devem ser confirmadas por um procedimento independente.
OBS 1: Nas pacientes femininas em que forem encontradas mutações patogênicas no gene STK11, mesmo que assintomáticas, a salpingo-ooforectomia redutora de risco é de cobertura obrigatória da mesma forma que a cobertura prevista para pacientes com diagnóstico de câncer, quando indicado pelo médico assistente. Da mesma forma, em todos pacientes de ambos sexos portadores de mutação patogênica de STK11, mesmo que assintomáticos, a colonoscopia e esofagoduodenoscopia periódicas são de cobertura obrigatória e devem ser realizadas conforme indicado pelo médico assistente.
110.43 – SÍNDROME DE POLIPOSE JUVENIL
1. Cobertura obrigatória de realização da análise genética para pacientes com pelo menos uma das seguintes características:
a. pelo menos 5 pólipos juvenis do colon e/ou reto confirmados por histologia;
b. qualquer número de pólipos juvenis quando localizados no trato gastro-intestinal excluindo-se colon e/ou reto confirmados por histologia;
c. qualquer número de pólipos juvenis quando localizados no trato gastro-intestinal confirmados por histologia em indivíduo com história familiar de síndrome de polipose juvenil.
2. Cobertura obrigatória para familiares de 1º, 2º, ou 3º grau de um indivíduo com mutação germinativa patogênica identificada nos genes BMPR1A ou SMAD4 (MADH4).
Método de análise genética utilizado de forma escalonada:
1. Nos casos em que a mutação genética já foi identificada na família, realizar apenas a pesquisa da mutação específica. Se houver na família uma mutação em SMAD4 o teste de um indivíduo em risco deve idealmente ser realizado nos primeiros 6 meses de vida devido ao risco de telangiectasia hemorrágica hereditária.
2. Nos casos de pacientes com suspeita clínica ou diagnóstico clínico conforme critérios acima, realizar:
a. Sequenciamento de Nova Geração do promotor, de toda região codificadora e das junções intron-exon dos genes BMPR1A e SMAD4. Se o Sequenciamento de Nova Geração não estiver disponível realizar Sequenciamento bidirecional do promotor, de toda região codificadora e das junções intron-exon por Sanger destes genes;
b. Em caso de resultado negati o no item “a” realizar MLPA de BMPR1A e SMAD4. Deleções de um éxon simples devem ser confirmadas por um procedimento independente.
OBS. 1: Nos pacientes em que forem encontradas mutações patogênicas nos genes BMPR1A ou SMAD4, mesmo que assintomáticos, a colonoscopia e esofagoduodenoscopia periódicas são de cobertura obrigatória e devem ser realizadas conforme indicado pelo médico assistente.
110.44 – RETINOBLASTOMA
1. Cobertura obrigatória da análise genética para pacientes de ambos os sexos com diagnóstico de retinoblastoma unilateral ou bilateral.
2. Cobertura obrigatória para pacientes de ambos os sexos com retinoblastoma e ao menos uma das seguintes características fenotípicas: atraso de desenvolvimento, retardo mental, microcefalia, coloboma, microftalmia, hipoplasia de polegar.
3. Cobertura obrigatória para aconselhamento genético e análise molecular de familiares de 1º, 2º e 3º graus de indivíduos com mutações identificadas em RB1.
Método de análise utilizado de forma escalonada:
I. Pacientes enquadrados no item 3 realizar apenas a análise da mutação específica identificada na família.
II. Pacientes enquadrados no item 1:
a. Sequenciamento de nova geração (NGS) no DNA constitutivo de toda a sequência codificadora e junções intron-éxon do gene RB1 ou alternativamente sequenciamento bidirecional de toda a sequência codificadora e junções intron-exon pelo método de Sanger em caso de indisponibilidade de NGS.
b. Em caso de resultado negati o em “a” realizar MLPA (Multi lex Ligation-dependent ProbeAmplification) para pesquisa de rearranjos gênicos em DNA constitutivo do gene RB1. Deleções de um éxon simples devem ser confirmadas por um procedimento independente,
c. Em caso de resultado negativo no ítem “a” ou “b” realzar sequenciamento de nova geração (NGS) no DNA tumoral (caso disponível) de toda a sequência codificadora e junções intron-exon do gene RB1 ou alternativamente sequenciamento bidirecional de toda a sequência codificadora e junções intron-exon pelo método de Sanger em caso de indisponibilidade de NGS.
III. Pacientes enquadrados no item 2:
a. Realizar o cariótipo com bandas GTG de alta resolução
b. Caso o paciente tenha cariótipo normal realizar o FISH ou aCGH para detecção de microdeleções submicroscópicas.
c. Caso o diagnóstico não tenha sido estabelecido através dos itens a e b realizar análise molecular conforme descrito no item II dos métodos diagnósticos escalonados deste subitem.
110.45 – SÍNDROME DE VON-HIPPEL-LINDAU
1. Cobertura obrigatória da análise genética para pacientes de ambos os sexos, que apresentem pelo menos duas das seguintes características:
a. Um ou mais hemangioblastomas da retina ou de cérebro
b. Diagnóstico de carcinoma de células claras renais
c. Feocromocitomas adrenais ou extra-adrenais
d. Tumores de saco endolinfático
e. Tumores neuroendócrinos do pâncreas.
f. Familiar de primeiro grau com história sugestiva de VHL
2. Cobertura obrigatória para o aconselhamento genético e análise molecular dos familiares de 1º, 2º ou 3º graus assintomáticos de um indivíduo com mutação do gene VHL.
Método de análise utilizado de forma escalonada:
1. Nos casos em que a mutação genética já tenha sido identificada na família, realizar apenas a pesquisa da mutação específica.
2. Nos casos que preenchem os critérios 1 e 2, realizar sequenciamento de Nova Geração de toda região codificante e junções intron-exon do gene VHL.
3. Quando não for possível realizar o Sequenciamento de Nova Geração, realizar o Sequenciamento por Sanger de toda região codificante e junções intron-exon do gene VHL.
4. Nos casos em que o diagnóstico não for estabelecido através dos itens acima, realizar MLPA (Multiplex Ligation-dependent Probe Amplification) para o gene VHL. Deleções de um éxon simples devem ser confirmadas por um procedimento independente.
110.46 – SÍNDROME DE GORLIN (SÍNDROME DE CARCINOMAS BASOCELULARES NEVÓIDES)
1. Cobertura obrigatória para pacientes que preenchem pelo menos:
a. dois (2) dos critérios maiores e um (1) dos critérios menores ou
b. um (1) dos critérios maiores e três (3) dos critérios menores.
Critérios Maiores:
a. Calcificação lamelar da foice;
b. Ceratocistos odontogênicos (com confirmação histopatológica);
c. Pits Palmares e/ou plantares (dois ou mais);
d. Carcinoma basocelular único diagnosticado antes dos 30 anos ou múltiplos carcinomas basocelulares (>5 ao longo da vida);
e. Familiar de 1º grau com de Síndrome de Gorlin.
Critérios Menores:
a. Meduloblastoma na infância (tumor primitivo do neuroectoderma – PNET);
b. Cistos pleurais ou linfomesentéricos;
c. Macrocefalia (perímetro cefálico acima do percentil 97 para idade);
d. Fenda labial e/ou palatina;
e. Anormalidade em costelas ou vertebras (costela extra ou bífida, vertebra bífida);
f. Polidactilia pré- ou pósaxial;
g. Fibromas cardiacos ou ovarianos;
h. Anomalias oculares (catarata, alterações pigmentares do epitélio da retina, defeitos de desenvolvimento como estrabismo, cisto orbital, microftalmia, hipertelorismo).
2. Cobertura obrigatória para realização do teste genético e aconselhamento genético para familiares de 1º, 2º, ou 3º grau de um indivíduo com mutação germinativa patogênica identificada no gene PTCH1.
Método de análise utilizado de forma escalonada:
1. Nos casos em que a mutação genética já foi identificada na família, realizar apenas a pesquisa da mutação específica.
2. Nos casos de pacientes com suspeita clínica ou diagnóstico clínico conforme critérios acima, realizar sequenciamento de Nova Geração (NGS) de toda região codificadora e junções intron-exon de PTCH1.
3. Quando não for possível realizar o Sequenciamento de Nova Geração, realizar o Sequenciamento por Sanger de toda região codificante e junções intron-exon do gene PTCH1.
4. Nos casos em que o diagnóstico não for estabelecido através dos itens acima, realizar MLPA (Multiplex Ligation-dependent Probe Amplification) para o gene PTCH1. Deleções de um éxon simples devem ser confirmadas por um procedimento independente.
110.47 – ANEMIA DE FANCONI – SÍNDROME COM INSTABILIDADE CROMOSSÔMICA ASSOCIADA A APLASIA MEDULAR, DEFEITOS CONGÊNITOS E CÂNCER
1. Cobertura obrigatória da análise molecular para pacientes com suspeita de Anemia de Fanconi (AF) definida pela presença de pelo menos um dos seguintes critérios:
a. Defeitos congênitos, especialmente do sistema esquelético redominantemente do eixo radial associado ou n o a manchas do tipo “café-com-leite” e déficit ondero-estatural;
b. Hipoplasia e/ou Aplasia da Medula Óssea envolvendo as três linhagens: hemácias, plaquetas e leucócitos; na presença ou não dos defeitos congênitos;
c. Síndrome Mielodisplásica associada aos defeitos congênitos;
d. Câncer embrionário infantil (meduloblastoma, neuroblastoma, tumor de Wilms) e/ou leucemia mielóide aguda associado aos defeitos congênitos;
e. Carcinoma de células escamosas de cabeça, pescoço e região genital associado aos defeitos congênitos.
2. Cobertura obrigatória para realização do teste genético e aconselhamento genético para familiares de 1o, 2o, ou 3o grau de um indivíduo com mutação germinativa patogênica identificada em um dos genes relacionados a anemia de Fanconi.
Método de análise molecular de forma escalonada e conforme cenários clínicos específicos:
Nos casos enquadrados no item 2 em que a mutação já foi identificada na família, realizar apenas a pesquisa da mutação específica.
1. Realizar teste citogenético utilizando as drogas Mitomicina C (MMC) ou Diepoxibutano (DEB) em culturas de linfócitos testando presença de instabilidade cromossômica DEB- ou MMC-induzida.
2. Se o item anterior for positivo e for necessária a definição do gene envolvido para fins de aconselhamento genético, realizar o Sequenciamento de Nova Geração envolvendo os genes FANCA, FANCG, FANCC, FACND2, FANCB, FACD1 (BRCA2), FANCN (PALB2), FANCO (RAD51C) e FANCP (SLX4).
OBS. 1: A identificação de determinadas mutações em heterozigose (BRCA2, PALB2, RAD51C e SLX4) aumenta a predisposição para câncer de mama e/ou ovário, portanto estes indivíduos devem ser encaminhados para manejo de alto risco de câncer.
